― 既知の7つのCO₂固定経路を網羅、未利用炭素資源の活用や地球規模の炭素循環の解明に貢献 ―
■ 概要
情報・システム研究機構 国立遺伝学研究所、同 データサイエンス共同利用基盤施設 ライフサイエンス統合データベースセンター(DBCLS)、独立行政法人製品評価技術基盤機構(NITE)、株式会社OKBPらの共同研究グループは、ゲノム情報から化学合成独立栄養細菌が持つ二酸化炭素(CO₂)固定経路を高精度に予測するソフトウェア「AutoFixMark(オートフィックスマーク)」を開発しました。また、本ツールの開発にあたり、既知の7つのCO₂固定経路すべてについて特徴的なマーカー遺伝子を定義しました。さらに、347株の化学合成独立栄養細菌のゲノム情報と保有するCO₂固定経路の情報を整備した高品質な参照データセットを構築しました。本研究成果により、メタゲノムやシングルセルゲノム解析などで得られる膨大な微生物ゲノムデータから、CO₂を資源として利用できる微生物を効率的に探索することが可能となり、地球規模の炭素循環の理解や、バイオテクノロジーを活用した持続可能なものづくりへの貢献が期待されます。本研究成果は、国際的な科学データ誌「Scientific Data」に2026年2月11日に掲載されました。
■ 成果掲載誌
・雑誌名 : Scientific Data
・論文タイトル : A curated resource of chemolithoautotrophic genomes and marker genes for CO₂ fixation pathway prediction
・論文タイトル(日本語) : CO₂固定経路予測のための化学合成独立栄養細菌ゲノムおよびマーカー遺伝子のキュレーション済みリソース
・著者 : Shuichi Kawashima, Yoko Okabeppu, Seiha Miyazawa, Natsuko Ichikawa, Hikaru Nagazumi, Yutaka Nishihara, Takeru Nakazato, Susumu Goto, Ken Kurokawa, Masaharu Ishii, Hiroshi Mori
・DOI : [
https://doi.org/10.1038/s41597-026-06655-z]
・URL :
https://www.nature.com/articles/s41597-026-06655-z
・AutoFixMarkソフトウェア:
https://github.com/h-mori/AutoFixMark
■ 研究の詳細
【研究の背景】
微生物によるCO₂固定は、炭素が制限された環境で微生物が生息するために不可欠なプロセスであり、地球全体の炭素循環において重要な役割を果たしています。化学合成独立栄養細菌が持つCO₂固定経路は多様で、カルビン・ベンソン回路(CBB回路)をはじめとする7種類が知られています。
しかし、これらの経路に関わる酵素遺伝子は多様な系統が所持しており、一部の酵素は複数の経路に関わっているため、ゲノム情報だけでどの経路を持っているかを正確に推定することは困難でした。細菌用の既存の代謝経路予測ツール(METABOLICやgapseqなど)は一般的な代謝経路の予測には有用ですが、多様なCO₂固定経路、特に比較的最近発見されたいくつかの経路の予測では、精度に課題がありました。
【研究の成果】
本研究グループは、以下の3つの成果を通じてこれらの課題を解決しました。
1.経路特異的なマーカー遺伝子の定義と予測ルールの構築
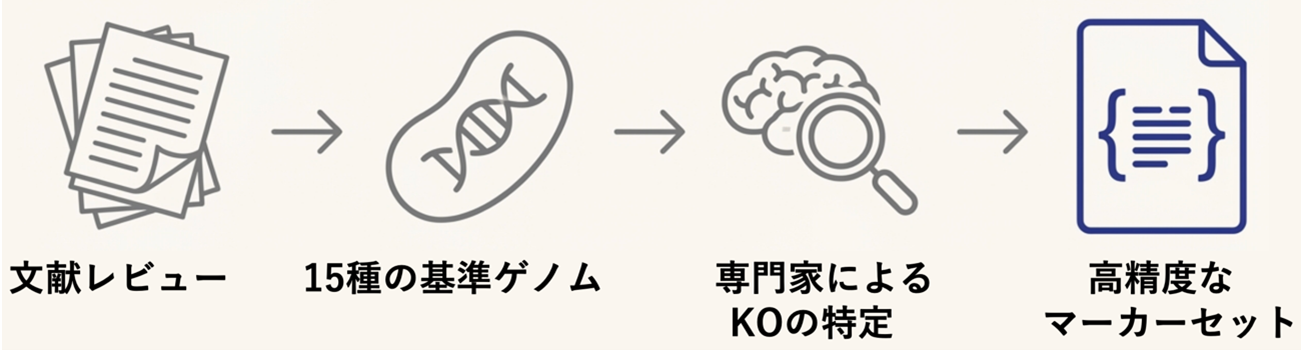
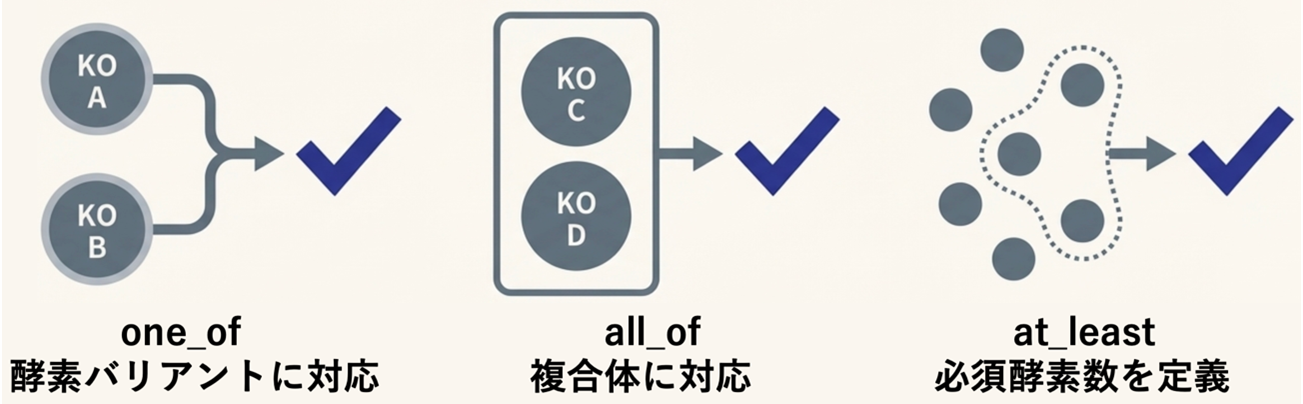
15種の代表的な化学合成独立栄養細菌のゲノム情報と文献情報を基に、既知の7つのCO₂固定経路すべてについて、経路の存在を特定するために不可欠な「マーカー酵素」とそれに対応する遺伝子のKEGG Orthology(KO)IDを定義しました(図1)。酵素の多様性に対応するため、「この遺伝子群のうち少なくとも1つがあればよい(one_of)」や「複合体を作るすべての遺伝子が必要(all_of)」といった柔軟な論理ルールを策定しました(図2)。
2.予測ツール「AutoFixMark」の開発
定義したマーカー酵素とルールに基づき、ゲノムの遺伝子リスト(KO組成)からCO₂固定経路の有無を自動判定するツール「AutoFixMark」を開発しました。このツールはPythonで動作し、GitHubレポジトリで公開済みであり誰でも無償で利用可能です。
3.高品質なベンチマークデータセットの構築と性能評価
ツールの性能を正しく評価するために、文献調査に基づいてCO₂固定能力が確認されている347株の微生物(16門を含む)のゲノム情報を、NITEグループが主体となって手動でキュレーション(精査)し、参照データセットを構築しました。 このデータセットを正解データとして、「AutoFixMark」と既存ツール(METABOLIC、gapseq)の予測精度を評価したところ、「AutoFixMark」は既存ツールでは予測が困難だった「ジカルボン酸/4-ヒドロキシ酪酸(DC/4HB)回路」や「還元的グリシン(rGly)経路」などの予測においても、高い予測精度を示しました。
図1. AutoFixMarkにおけるCO₂固定経路のマーカー酵素の定義方法
図2. 各経路の酵素所持のルールを定義
【今後の展望】
「AutoFixMark」は、ゲノム配列さえあればCO₂固定経路の有無を予測できるため、培養が困難な微生物を含むメタゲノム解析データやシングルセルゲノムへの適用が容易です。本ツールは、環境中における独立栄養細菌の系統的な多様性の解明に役立つだけでなく、CO₂を原料として有用物質を生産する微生物の探索など、脱炭素社会の実現に向けたバイオテクノロジーの応用への基盤となることが期待されます。
すべてのキュレーション済み酵素遺伝子セット、予測ルール、AutoFixMarkソフトウェア、およびベンチマークデータセットは、GitHubレポジトリおよびデータレポジトリZenodoを通じて公開済みであり、世界中の研究者が利用可能なリソースとなっています。
■ 用語解説
化学合成独立栄養細菌(Chemolithoautotrophs)
光エネルギーではなく、水素や硫黄化合物などの無機物の化学エネルギーを利用して、CO₂を有機物に変換(固定)して増殖できる細菌のこと。
KEGG Orthology(KO)
遺伝子やタンパク質の機能を分類したデータベース。生物種を超えて共通する機能を持つ遺伝子に同じKO IDが割り振られるため、ゲノム解析において機能推定の基準として広く利用されている。京都大学化学研究所の金久研究室で開発され運用されているデータベースである。
7つのCO₂固定経路
これまでに自然界で知られている化学合成独立栄養細菌が持つCO₂固定経路は以下の7経路である。
・カルビン・ベンソン・バッシャム(CBB)回路
・還元的トリカルボン酸(rTCA)回路
・ウッド・リュングダール(WL)経路
・3-ヒドロキシプロピオン酸(3HP)回路
・3-ヒドロキシプロピオン酸/4-ヒドロキシ酪酸(3HP/4HB)回路
・ジカルボン酸/4-ヒドロキシ酪酸(DC/4HB)回路
・還元的グリシン(rGly)経路
■ 研究体制と支援
本研究は主に、国立研究開発法人新エネルギー・産業技術総合開発機構(NEDO)のグリーンイノベーション基金事業(JPNP22010)、および国立研究開発法人科学技術振興機構(JST)NBDC事業推進室の統合化推進プログラム(JPMJND2206)の支援を受けて行われました。